【期刊信息】
Message刊名:计算机光盘软件与应用
主办:中国大恒公司大恒电子出版社
主管:中国科学院
ISSN:1007-9599
CN:11-3907/TP
语言:中文
周期:半月
影响因子:0
期刊分类:计算机软件及计算机应用
用计算机真正模拟生命还要多久
作者:网站采编
关键词:
导语
当前在计算科学、计算机硬件和生命科学方面无法预料的突破可能会使我们朝着目标迈进计算机模拟生物学的速度更快。
Roland R. Netz、William A. Eaton?| 作者赵雨亭?| 译者
刘培源?|?审校
邓一雪|?编辑

论文题目:
Estimating computational limits on theoretical descriptions of biological cells
论文地址:
埃尔温·薛定谔在他1944年的著作《生命是什么?》[1]中问道:“如何使用物理兼化学方法来解释在一个活的有机体在自己的空间边界内发生的时空事件?”在随后的近80年中,科研人员已经探索了许多有关亚细胞现象的、更深层机理的成果。虽然如此,以第一原理对生活中的生命体进行计算机模拟仍然是一个遥不可及的目标。
在最近一篇发表于美国国家科学院院刊 PNAS 的文章中[2],Netz 与 Eaton 预测了这种模拟变为现实所需的时间。他们进一步将其估计扩展到更大的生物系统,并讨论是否以及何时有可能模拟诸如人脑之类的多细胞实体。
1. 用计算机模拟生命过程
在一个生物学上有意义的时间间隔内(例如一个小时、一次细胞分裂时间),模拟细胞大小的分子装配似乎是一项艰巨的任务。的确,现代分子模拟领域的胜利之一就是观察到了蛋白质的可逆老化——这种老化发生在微秒级[3]。相比之下,单个细胞每小时可以合成数千或数百万个蛋白质,其中许多蛋白质可能需要数秒或数分钟才能折叠,并且只有在细胞机械(cellular machinery)的帮助下才能完成。
在最新的科研尝试中[4],研究者模拟了细胞质中,相对较大的100 nm×100 nm×100 nm亚空间。但是,该研究中获得的数十纳秒的时间尺度却非常短,比所需的1小时短了10个数量级!但是,如果按照摩尔定律的预测,计算能力继续呈指数增长,那么弥合这种时间尺度上的差距似乎并非遥不可及。
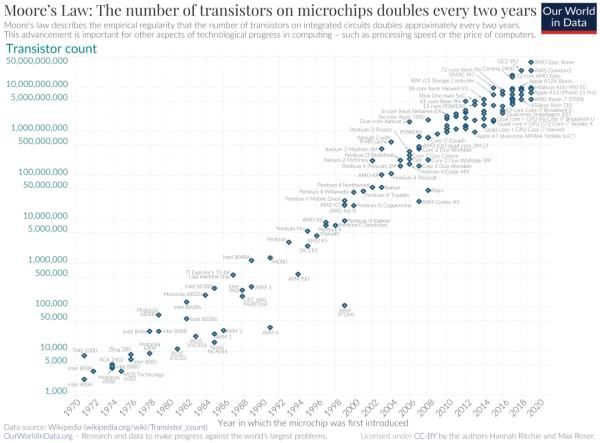
图1. 摩尔定律认为集成电路可容纳的晶体管数量,每隔约18个月翻倍,代表着对计算能力指数增长的观测和预期。但随着芯片组逐渐接近单个原子尺度,摩尔定律可能会失效。
的确,如果计算机速度每 1.5 年翻一番,则应该有可能在50年[5]内达到所需的时间范围。某些为加速分子模拟而开发的方法可能会提供进一步的帮助。例如,可以尝试提高仿真温度以加快动力学速度。尽管这种简单的加速动力学的方法存在问题——比如我们无法通过将葡萄酒在不破坏质量的前提下升温来加速老化——但该思路仍然具有启发。
然而,存在一个关键问题:分子动力学(Molecular dynamics,MD)作为标准在原子级上对生物分子现象进行建模的方法并不是真正的第一手方法:要根据第一性原理方法(first-principles method)预测分子间与分子间相互作用,必须为电子和原子核求解量子薛定谔方程(Schr?dinger equation)——这需要巨大算力。
2. 简化模拟降低算力
从头开始使用分子动力学法求解的代价就是可观的计算成本。相比之下,标准的分子动力学方法利用了两种简化方法:1)由于电子的移动速度比原子核快得多,因此它们可以使原子核感受到有效的相互作用;2)可以使用经验“力场(force field)”来近似这种有效的相互作用,但它只是描述有效势能随核坐标的函数而变化的分析公式。经过数十年的蓬勃发展,现在力场已非常精确且可靠。不幸的是,当前使用的大多数力场仍然存在基本局限性:科研人员无法对其进行化学处理。
化学反应涉及共价键和断裂和形成。没有化学反应,就不会有生命。细胞内的酶催化许多化学反应,包括与代谢过程或机械运动产生有关的化学反应。为了描述这种反应,必须进行量子处理。Netz 和 Eaton 指出:幸运的是,不需要量子力学来描述整个单元。相反,研究人员仅需要量子力学处理来描述形成的原子的有限子集——如酶的活性位点及其底物。
细胞模拟的量子部分值得进一步研究。化学反应的速率(即每单位时间发生反应的概率)通常可以由阿伦尼乌斯定律(Arrhenius law)来描述,
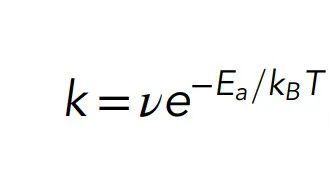
其中ν是一个前置因子,kBT是热能(等于玻尔兹曼常数和温度的乘积),Ea是活化能,可以粗略地解释为阈值能量。在反应中,必须提供给该分子活化能Ea才能使其从稳定的分子构象中消失,从而过渡到反应产物(图2)。如果T是人体的温度,则有kBT≈0:6kcal = mol。
文章来源:《计算机光盘软件与应用》 网址: http://www.jsjgprjyyy.cn/zonghexinwen/2021/0331/892.html
投稿电话: 投稿邮箱:



